Forschungsexpedition in den Zellkern
Was steuert unsere Gene? Welche Mechanismen sorgen dafür, dass sie im richtigen Moment ein- oder ausgeschaltet werden? Darauf sucht die Chromatinbiologin Raffaella Santoro von der Universität Zürich mit ihrem ERC-Projekt Antworten. Protokoll einer Begegnung mit einer Grundlagenforscherin.
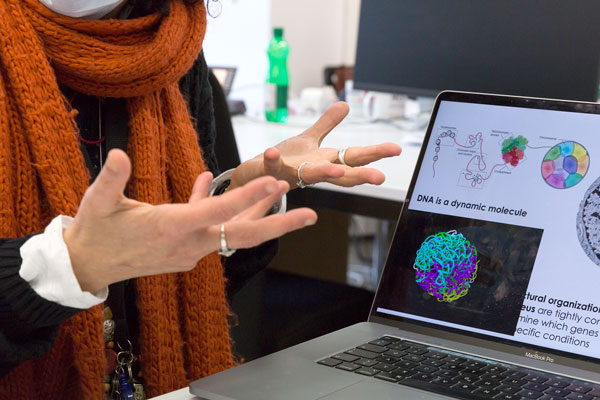
Raffaella Santoro greift zu einer Schnur und ballt sie zu einem Bündel. «Sehen Sie, etwa so sieht ein Chromosom im Kern unserer Zellen aus. Unsere Erbinformation, die DNA, ist auf kompakt gezwirnten Chromatinschnüren aufgereiht. Damit diese Schnüre im winzigen Zellkern Platz finden, falten sie sich nochmals zu komplexen Schnurbündeln. Dies geschieht nicht zufällig. Die Struktur, die diese Bündel haben und der Ort, an dem sie im Zellkern platziert sind, spielen eine wichtige Rolle bei der Regulation der Gene. Aber was da im Detail abläuft, verstehen wir noch nicht genau und das versuche ich herauszufinden.»
Wir treffen Raffaella Santoro im Büro ihres Labors auf dem Campus Irchel der Universität Zürich. Durch das grosse Fenster scheint hell die Wintersonne. Wir haben die renommierte Chromatinbiologin gebeten, uns ihr ERC-Forschungsprojekt zu erklären, mit dem sie herausfinden will, was die Gene im Zellkern steuert und welche Rolle die Chromosomenstruktur dabei spielt. Nun sitzt sie uns an einem Tisch gegenüber und öffnet meinem Fotografen und mir an diesem Nachmittag mit grosser Geduld und italienischem Charme ein Fenster in die faszinierende Welt ihrer biologischen Grundlagenforschung.
Was Wollfäden in einer Plastikschüssel erzählen
Raffaella Santoro legt das Schnurbündel zur Seite und stellt eine Plastikschüssel auf den Tisch. Darin sind Knäuel bunter Wollfäden um einen schwabbeligen Ballon gruppiert, in dem sich etwas Wasser befindet. «Um Ihnen meine Forschung zu erklären, habe ich für Sie dieses einfache Modell des Zellkerns gebastelt», sagt sie lachend und zeigt auf den Schüsselrand. «Die Plastikschüssel steht für die Zellkernhülle. Sie ist fest und hat auf der Innen- und der Aussenseite je eine Membran, die den Zellkern vom Zytoplasma trennt. Die bunten Wollknäuel symbolisieren die verschiedenen Chromosomen, die an ganz bestimmten Stellen im Kern platziert sind. Der schwabbelige Ballon, der hier zwischen den Knäueln liegt, steht für das kleine Kernkörperchen, den Nukleolus, das sich in jedem Zellkern findet. Seine Form ist flexibel, wie ein Öltropfen im Wasser. Es hat auch keine feste Wand und keine Membran. Aber dieses unscheinbare Ding spielt eine Schlüsselrolle bei den Steuerungsprozessen des Lebens. Ohne den Nukleolus gäbe es uns wohl gar nicht.» Mit Hilfe dieses bunten Zellmodells erklärt uns die Spitzenforscherin in der folgenden Stunde anschaulich, wonach sie sucht und wie sie arbeitet.
Solange wir leben sind die Zellen unseres Körpers «im Dauerbetrieb». Ständig werden Proteine hergestellt, umgebaut, abgebaut und weitertransportiert. Die gesamten «Baupläne» dieser komplexen Prozesse sind in der DNA festgehalten, die sich auf den Chromosomenschnüren befindet. Dort werden die benötigten «Baupläne», die Gene, bei Bedarf abgelesen, als mRNA kopiert und ins Zytoplasma der Zellen transportiert, wo sie die programmierten Prozesse in Gang setzen. Wie aber «weiss» die Zelle, wann welche Gene wie lange eingeschaltet werden müssen und welche nicht? Wie funktioniert dieser Steuerungsmechanismus, der dafür sorgt, dass die «richtigen» Abschnitte der DNA zum «richtigen» Zeitpunkt aktiviert oder deaktiviert werden? Raffaella Santoro hat mit ihrem ERC-Forschungsprojekt dazu wichtige neue Erkenntnisse gewonnen.
«Sehen Sie», sagt sie und zeigt auf einen der bunten Wollfäden ihres Zellkernmodells, «wenn die Gene aktiviert sind, befindet sich der Abschnitt des Chromosoms, auf dem die eingeschalteten Gene liegen, meist hier, in der Mitte des Knäuels. Wenn die Gene inaktiv sind, finden wir den Chromosomenabschnitt entweder in der Nähe der Zellkernhülle oder direkt am Nucleolus. Wir haben nun herausgefunden, dass die meisten DNA-Abschnitte, deren Gene ausgeschaltet sind, den Nucleolus berühren, aber jene Gene, die aktiv sind, den Nucleolus nie touchieren. Dieser Mechanismus hat zur Folge, dass die Chromosomenfadenknäuel ihre Struktur immer wieder verändern, je nachdem, welche Gene gerade aktiviert oder deaktiviert sind. Wenn wir diese ständigen Strukturveränderungen der Chromosomenbündel in Momentaufnahmen festhalten, entsteht eine dynamische dreidimensionale Karte der komplexen Genomarchitektur im Zellkern, die je nachdem, welche Gene gerade ein- oder abgeschaltet sind, anders aussieht. Die momentane Genomarchitektur spiegelt die momentanen Genaktivitäten. Und diese unterscheiden sich von Zelltyp zu Zelltyp. Wir haben etwa 200 verschiedene Arten von Zellen in unserem Körper. Sie enthalten alle dieselbe DNA. Aber je nachdem, ob es sich um eine Hirn-, Leber- oder Blutzelle handelt, werden nur jene Genabschnitte aktiviert, die für die Funktion dieses Zelltyps wichtig sind. Die anderen werden abgeschaltet.»
Der Trick mit dem Enzym
Wir haben Raffaella Santoros Erklärungen fasziniert zugehört und möchten nun wissen, wie sie feststellen kann, welche Genabschnitte den Nucleolus berühren? Dazu musste die Forscherin zuerst eine Methode entwickeln und an Stammzellen von Mäusen erproben und validieren, deren Epigenom und Genexpressionszustand bekannt ist. Der Knackpunkt war, einen Weg zu finden, über den sich die Gene, die den Nucleolus berühren, markieren lassen. Raffaella Santoro und ihrem Techniker Dominik Bär ist es gelungen, ein Enzym herzustellen, das sich am Nucleolus anbringen lässt. Berührt ein Gen den Nucleolus, setzt ihm das Enzym eine Art Stempel auf. Eine DNA-Analyse zeigt dann, um welches Gen es sich handelt. Im Abgleich mit dem bekannten Epigenom und Genexpressionszustand der Mäusestammzellen lässt sich hierauf feststellen, welche Funktionen die markierten Gene haben und ob sie aktiv oder inaktiv sind. Auf diese Weise hat Raffaella Santoros Forschungsteam Mäusestammzellen in unterschiedlichen Stadien ihrer Entwicklung untersucht und so ein umfassendes Bild ihrer dynamischen Genomstruktur erhalten.
Doch das ist erst der Anfang. Im zweiten Teil ihres Projekts hat die Biowissenschaftlerin nun begonnen, diese Methode auf Krebszellen anzuwenden. Dabei will sie herausfinden, welche Gene von Prostatakrebszellen den Nucleolus berühren und somit auf inaktiv geschaltet sind. Dann kommt Big Data ins Spiel. Die empirischen Resultate wird ihr Forschungsteam mit den vielen Daten aus der Prostatakrebsforschung und der Klinischen Forschung abgleichen, um zu sehen, ob die identifizierten Gene bei Prostatakrebs eine Rolle spielen.
Klein und unterschätzt
Zum Thema Prostatakrebs forscht Raffaella Santoro schon seit vielen Jahren. Ihre Methode eröffnet ihr nun völlig neue Möglichkeiten. «Wir haben gesehen, dass der Nucleolus von Prostatakrebszellen sehr viel grösser ist als jener normaler Prostatazellen. Wenn sich der Nucleolus vergrössert, verändert sich auch die Struktur im Zellkern. Mit unserer Methode können wir jetzt herausfinden, welche Gene von diesen strukturellen Veränderungen betroffen sind.» Damit legt die ambitionierte Wissenschaftlerin einen weiteren Trittstein auf dem langen Weg zum Verständnis der Mechanismen, die zu Prostatakrebs führen. Am Ende dieses Weges könnten vielleicht einmal neuartige Therapien entwickelt oder der Krebs gar verhindert werden.
Doch zurück zum Nucleolus. Das kleine Kernkörperchen hat die kreative Biochemikerin im Lauf ihrer Karriere immer wieder beschäftigt und die Initialzündung für ihr ERC-Projekt ausgelöst. «Der Nucleolus fasziniert mich seit meiner Zeit als Postdoktorandin am Deutschen Krebsforschungszentrum in Heidelberg vor zwanzig Jahren. Wir wissen, dass der Nucleolus eine Schlüsselrolle spielt bei der Regulierung der Lebensprozesse. Aber wir wissen noch immer wenig über seine vielen Funktionen. Dazu kommt, dass die Untersuchung seines Inhalts sehr komplex ist.» Über drei Jahre trieb Raffaella Santoro die Frage um, wie sie die Interaktion zwischen Genen und Nucleolus nachweisen könnte. Schliesslich hatte sie eine Idee. «Ich habe einen wunderbaren Techniker in meinem Lab. Ich ging also zu Dominik Bär und schilderte ihm, dass wir eine Methode brauchten, mit der wir die Gene, die den Nucleolus berühren, markieren könnten und dass ich eine Vorstellung hätte, wie dies funktionieren könnte. Wir führten ein paar Experimente durch, kreierten dieses Enzym und siehe da, es funktionierte!» Daraufhin schrieb sie sogleich einen ERC-Antrag und erhielt den Grant.
Dank ihres ERC-Projekts kann sich Raffaella Santoro nun intensiv mit der Funktion des Nucleolus auseinandersetzen. Dabei geht sie mit ihrem Team auch neue Wege. Eine ihrer Postdoktorandinnen hat sich darauf spezialisiert, den Nucleolus einer einzelnen Zelle mit einem Laser aufzuschneiden. Der Inhalt wird sequenziert und gibt neue Einblicke frei.
«Kann man denn sagen, dass der Nucleolus darüber entscheidet, welche Gene deaktiviert werden?», versuche ich das, was wir erfahren haben, zusammenzufassen. «Nein, das wäre dann doch zu einfach!», ruft Raffaella Santoro mit gespielter Empörung und lacht. «Wir wissen, dass die meisten Gene, die den Nucleolus touchieren, inaktiv sind. Aber wir wissen nicht, ob sie ausgeschaltet sind, weil sie den Nucleolus berühren oder ob sie den Nucleolus berühren, weil sie ausgeschaltet sind. Wir haben im Moment nur die Korrelation: Gene, die den Nucleolus berühren, sind inaktiv. Um zu verstehen, warum das so ist, müssen wir den Mechanismus dahinter kennen. Dazu müssen wir identifizieren, was ein Gen veranlasst, den Nucleolus zu touchieren und herausfinden, was mit einem inaktiven Gen geschieht, wenn es mit dem Nucleolus nicht mehr in Kontakt ist. Aber so weit sind wir noch nicht.»
«Und wie geht es weiter?», frage ich die passionierte Grundlagenforscherin am Ende dieses Nachmittags. «Wir werden uns alle Aspekte des Nucleolus in gesunden und kranken Zellen ansehen und vergleichen, bis wir die Mechanismen verstehen», antwortet sie fröhlich.
Interview mit Raffaella Santoro (in englisch)
Raffaella Santoro
Raffaella Santoro wuchs in Rom auf. Sie studierte an der Universität Rom La Sapienza Chemie und promovierte dort in Biochemie. Nach dem Doktorat ging sie 1994 ans Hans-Knöll-Institut in Jena, wo sie drei Jahre als Postdoktorandin arbeitete. Danach trat sie eine zweite Postdoktorandenstelle am Deutschen Krebsforschungszentrum in Heidelberg an. Im Jahr 2007 zog Raffaella Santoro mit ihrer Familie nach Zürich und wurde Leiterin einer Forschungsgruppe am Department of Biosystems Science and Engineering (D-BSSE) der ETH Zürich. Von dort wechselte sie 2009 an die Universität Zürich, wo sie am heutigen Department für Molekulare Mechanismen bei Krankheiten (DMMD) die Leitung einer Forschungsgruppe übernahm und 2019 zur Professorin für Stammzell- und Chromatinbiologie ernannt wurde. In ihrer Gruppe «Santoro Lab» arbeiten zurzeit 15 Forscherinnen und Forscher daran, die Grundprinzipien der Genregulierung zu verstehen.
Neben ihrer wissenschaftlichen Arbeit ist Raffaella Santoro seit einiger Zeit eine passionierte Köchin. Am liebsten kocht sie Gerichte aus Rom und Mittelitalien nach den überlieferten Rezepten ihrer Mutter und Grossmutter.
Horizon 2020 Projekt
NucleolusChromatin: Analysis of the nucleolus in genome organization and function
- Projektart: ERC Advanced Grant
- Laufzeit: 1. September 2018 – 31. August 2023 (60 Monate)
- Beitrag für die Universität Zürich: 2’500’000 €