Von Viren, Geflügel und Menschen
Wie die Epidemiologin Claire Guinat den Genen von Vogelgrippeviren das Geheimnis ihrer Verbreitung entlockt, wie sich dadurch die Geflügelpest besser bekämpfen lässt und wie dieses Wissen der Gesundheit von Menschen und Tieren dient.
An Ende dieser Geschichte geht es um Geflügel und Menschen. Aber sie beginnt mit zwei Vogelgrippeviren. Sie tragen die Bezeichnung H5N8 und H7N9 und haben Seuchengeschichte geschrieben. Das H5N8 Virus gelangte wahrscheinlich mit Zugvögeln nach Europa, infizierte die Geflügelbetriebe auf dem ganzen Kontinent und löste zerstörerische Epidemiewellen aus. Besonders schlimm wütete die Seuche im Winter 2016 und 2020, als in Hunderten von Betrieben Millionen von Hühnern, Enten und anderes Geflügel getötet werden mussten, um die Epidemie unter Kontrolle zu bringen. Menschen wurden vom H5N8 Virus bisher kaum angesteckt. Anders das H7N9 Virus, das vor allem in China unter dem Geflügel grassiert. Seit 2013 werden aus China Übertragungen des H7N9 Virus auf Menschen gemeldet, die schwer erkranken. Mit welchen Strategien lassen sich die Seuchenzüge dieser Vogelgrippeviren verhindern oder zumindest rasch eindämmen? Wie lassen sich Mensch und Tier vor Ansteckungen schützen? «Indem wir die Infektionsdynamik dieser Viren verstehen», antwortet die junge Veterinär-Epidemiologin Claire Guinat, als wir sie am ETH Departement für Biosysteme (D-BSSE) in Basel zum Gespräch treffen.
Forschen zwischen Basel und Toulouse
Schon nach dem ersten Studienjahr an der Ecole Nationale Vétérinaire de Toulouse (ENVT) war der Veterinärstudentin Claire Guinat klar: Sie wollte in die Forschung. «Mich interessierten die Infektionskrankheiten bei Nutztieren wie Hühner, Schweine und Rinder.
«Schon nach dem ersten
Studienjahr war mir klar:
Ich wollte in die Forschung.»
Ich wollte wissen, wie sich diese verbreiten und wie sie sich bekämpfen lassen», erzählt uns die vife Französin im Kaffeeraum der Forschungsgruppe für Computational Evolution cEvo, in der sie als Marie Skłodowska-Curie Fellow zurzeit arbeitet. Also spezialisierte sie sich im Masterstudium auf Epidemiologie infektiöser Krankheiten von Tieren, schrieb am Royal Veterinary College in London (RVC) und dem Pirbright Institute eine Doktorarbeit zur Übertragung des Afrikanischen Schweinepestvirus und trat 2016 eine Stelle in Toulouse als Postdoc an der ENVT und dem Nationalen Forschungsinstitut für Landwirtschaft, Nahrung und Umwelt (INRAE) an. Es war die Zeit der grossen Hühnerpestepidemie in Europa und Claire Guinat erlebte die Auswirkungen der Seuche hautnah, sowohl bei ihren Kontakten mit den Geflügelhaltern wie auch in ihren Forschungsprojekten. Durch die Analyse epidemiologischer Daten wie Standorte der infizierten Betriebe, Zeitpunkt des Seuchenausbruchs, Anzahl der Tiere je Betrieb usw. versuchte sie zu erkennen, wie sich die Seuche verbreitet. Mit mathematischen Modellierungen ermittelte sie die Faktoren, welche die Verbreitung der Krankheit zwischen den Geflügelbetrieben vorantrieben. «Das waren klassische epidemiologische Forschungsansätze, mit denen ich damals und in den Jahren danach arbeitete. Aber ich wollte meine wissenschaftlichen Fähigkeiten und Kenntnisse erweitern und ich wollte lernen, epidemiologische Daten mit genetischen Daten zu kombinieren, um so zu präziseren Analysen zu gelangen. Also meldete ich mich 2018 zum Workshop «Taming the Beast» an, den Tanja Stadlers Forschungsgruppe cEvo jeweils im Sommer anbietet», schildert Claire Guinat. Tanja Stadler, Professorin für Computational Evolution an der ETH Zürich, ist bekannt für ihre Arbeiten zur Phylogenetik und Phylodynamik; zwei zentrale Gebiete der modernen Biologie. Mit Hilfe der Phylogenetik lassen sich die evolutionären Beziehungen von Organismen wie in einem «Familienstammbaum» rekonstruieren. Die Dynamik, in der diese Beziehungen ablaufen, enthüllt die Phylodynamik, die auf anspruchsvollen mathematischen Modellierungen beruht. In einwöchigen Sommerkursen vermitteln Tanja Stadler und ihr Team jungen Forschenden, wie sie diese Ansätze auf ihre eigenen Gendatensätze anwenden und so neue Informationen gewinnen können.
«Ich wollte lernen,
epidemiologische Daten
mit genetischen Daten
zu kombinieren.»
Am Ende des Kurses war für Claire Guinat klar – sie wollte diese neuen Methoden, die im Gebiet der Tiergesundheit noch kaum bekannt waren, lernen und sie künftig in ihren Forschungsprojekten anwenden. Sie fragte Tanja Stadler, ob sie diese Kompetenz mit einem Projekt bei ihr in Basel erwerben könne und Tanja Stadler sicherte ihre volle Unterstützung zu. Zurück in Toulouse begann Claire Guinat sogleich, den Antrag für ihr Forschungsprojekt DIGDEEP zu schreiben. Mit dem phylodynamischen Ansatz sollten die genetischen Daten der H5N8 und der H7N9 Vogelgrippeviren, die von 2013 bis 2020 in Europa und Asien grassierten, ausgewertet und mit den epidemiologischen Daten kombiniert werden mit dem Ziel, neue Erkenntnisse über die Verbreitungsmechanismen dieser Viren zu gewinnen. Daraus liessen sich dann neue Strategien und Massnahmen zu Prävention und Bekämpfung künftiger Tierseuchen entwickeln. Mit diesem Projekt bewarb sich Claire Guinat um ein Marie Skłodowska-Curie Stipendium und erhielt die Zusage.
Im September 2020 startete sie ihr Projekt als Marie Skłodowska-Curie Fellow in Tanja Stadlers Forschungsgruppe cEvo am ETH Departement BSSE in Basel. Seither pendelt sie zwischen Basel und Toulouse, da die ENVT als Partnerinstitution mit im Boot ist. «Hier in Basel lerne ich, wie sich die Übertragung von Viren mit phylodynamischen Instrumenten quantifizieren lässt. Die Kolleginnen und Kollegen zeigen mir, wie die phylodynamischen Modelle funktionieren, welche Mathematik dahintersteckt, welche Möglichkeiten die Tools bieten, wie sie anzuwenden sind und wie ich sie auf meine spezifischen Fragen anpassen kann. In Toulouse lerne ich alles über die Vogelgrippeviren, ihre Struktur, ihre Mutationen, ihre Verbreitungswege», beschreibt Claire Guinat ihr Fellowship.
Die Übertragungsdynamik quantifizieren
Im Verlauf einer Epidemie wie der Hühnerpest mutiert das Virus in kurzer Zeit immer wieder, sodass im Geflügelbetrieb Z, der zuletzt infiziert wurde, eine mehrfach mutierte Virusvariante jenes Erregers auftritt, mit dem die Infektionswelle im Geflügelbetrieb A begann. Erkennen und unterscheiden lassen sich die verschiedenen «Virus-Generationen» durch die Sequenzierung der Genome der Virusproben, die in den infizierten Betrieben gesammelt werden. Da aus den epidemiologischen Daten bekannt ist, wann in welchen Betrieben die Seuche ausgebrochen ist, können die Mutationen des Virus im Zeitverlauf zurückverfolgt werden. Mit diesen Informationen lässt sich ein phylogenetischer Baum aus den gesammelten Virusproben skizzieren, der ihre «Abstammungslinien» zeigt. Er gleicht einem Familienstammbaum, der den Urahn und die nachfolgenden Generationen abbildet und erkennen lässt, wer mit wem wie verwandt ist. Der phylogenetische Baum zeigt aber nicht nur, wie unterschiedlich die Virusproben sind. Er vermittelt auch ein Gesamtbild des Verbreitungsprozesses der Epidemie. Da das Virus nach bestimmten evolutionären Mustern mutiert, kann der Baum auch dazu verwendet werden, Viruslinien abzuleiten, für die keine Proben gesammelt wurden. Dies bedeutet, dass anhand des Baumes auf die Anzahl Betriebe geschlossen werden kann, die während der Epidemie möglicherweise unentdeckt blieben und bei denen daher keine Proben entnommen wurden. Doch dies ist nur der erste Schritt der Analyse. Im zweiten werden mit Hilfe der Phylodynamik die Parameter berechnet und quantifiziert, welche für die Übertragungsdynamik einer Epidemie bestimmend sind. Claire Guinat beschreibt dies so: «Wir können dann beispielsweise sagen, dass die Epidemie vor allem durch lokale Ansteckungen von Geflügelbetrieb zu Geflügelbetrieb und nicht durch Wildvögel befeuert wurde. Aus solchen Erkenntnissen lassen sich dann Präventions- und Bekämpfungsstrategien ableiten.»
Phylogenetik und Phylodynamik – die Schlüsselwerkzeuge
Claire Guinat führt uns an ihren Arbeitsplatz, wo sie uns zeigt, wie sie konkret arbeitet. In den ersten Monaten ihres Fellowship trug sie tausende von Genomsequenzierungsdaten des H5N8 und H7N9 zusammen, die während der Epidemien von Forschungsinstituten in Europa und Asien erhoben wurden.
«Mein Traum wäre, dereinst
eine eigene international
zusammengesetzte Forschungsgruppe
zu leiten.»
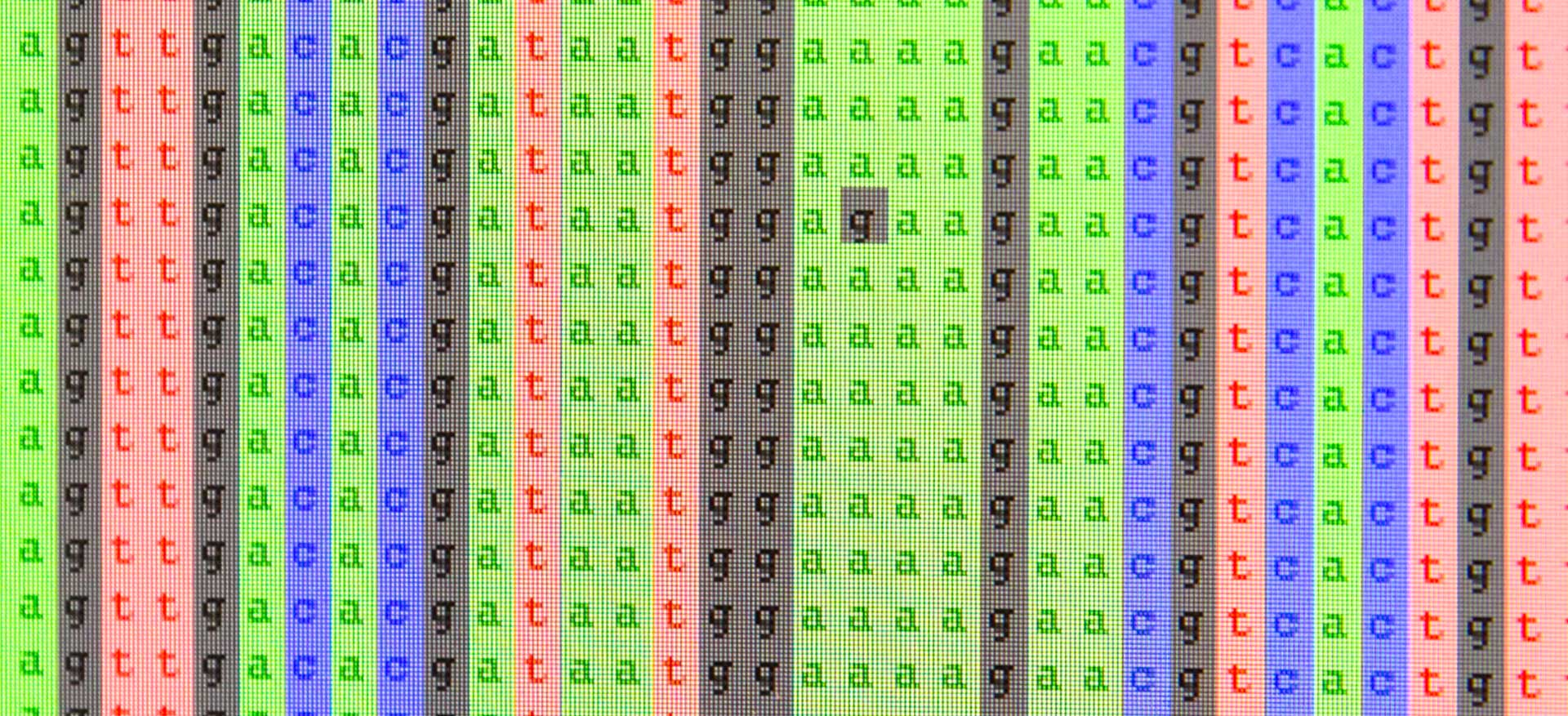
Auf dem Bildschirm ihres Laptops erscheint eine lange Tabelle mit Hunderten von Zeilen. Jede Zeile steht für eine Genomsequenz des H5N8 bzw. des H7N9 Virus und enthält Angaben zu Ort und Datum der Probeentnahme. Jede dieser Sequenzen besteht aus einer Buchstabenfolge und jeder Buchstabe steht für ein Nukleotid. Alle diese Sequenzen sind so ausgerichtet, dass ähnliche Nukleotide an einer bestimmten Stelle der Buchstabenfolge dieselbe Farbe tragen, wodurch die Veränderungen der Genome Zeile für Zeile sofort zu erkennen sind. Mit diesen Daten erstellt Claire Guinat phylogenetische Stammbäume, aus denen sie dann mit Hilfe der phylodynamischen Tools die Parameter abschätzt, die für die Ausbreitung der Krankheit entscheidend sind. Die Forschungsarbeit zum H5N8 hat Claire Guinat bereits weitgehend abgeschlossen und konzentriert sich jetzt, in der zweiten Hälfte ihres Projekts, auf die Auswertung der Daten zum H7N9 Virus. Am 31. August 2022 endet ihr Fellowship. Welche Pläne hat sie für ihre Zukunft? Claire Guinat lacht. «Mein Traum wäre, dereinst eine eigene international zusammengesetzte Gruppe zu leiten, die sich mit Epidemien von Tieren beschäftigt und dazu verschiedene Methoden verwendet. Und da viele Infektionskrankheiten von Tieren ja irgendwann auf Menschen übergehen dient diese Forschung am Ende auch der Gesundheit der Menschen.»
Interview mit Claire Guinat (englisch)
Claire Guinat
Claire Guinat studierte Veterinärmedizin an der Ecole Nationale Vétérinaire Toulouse (ENVT) und schloss 2013 mit einem Master in Veterinär-Epidemiologie ab. Danach war sie von 2013 - 2016 am Royal Veterinary College (RVC) und am Pirbright Institute in Grossbritannien, wo sie 2016 in Veterinär-Epidemiologie doktorierte. Zurück in Frankreich erhielt sie eine Forschungsstelle als Postdoktorandin an der Ecole Nationale Vétérinaire Toulouse und dem Institut National de Recherche pour l'Agriculture, l'Alimentation et l'Environnement (INRAE).
Im September 2020 startete Claire Guinat als Marie Skłodowska-Curie Fellow ihr Forschungsprojekt DIGDEEP (Digging deeper into genes to track infectious disease outbreaks) in der cEvo-Forschungsgruppe am Departement für Biosysteme (D-BSSE) der ETH Zürich in Basel. Sie fokussiert in ihrer Forschung darauf, die Ausbreitung von Infektionskrankheiten zu verstehen und zu quantifizieren mit dem Ziel, Tiergesundheit zu fördern und zu erhalten. In ihrer Freizeit läuft Claire Guinat Mittel- und Langstrecken, wandert und macht Yoga. Aber am liebsten verbringt sie die Zeit mit ihrem Partner und ihren beiden kleinen Söhnen.
Horizon 2020 Projekt
DIGDEEP: Digging deeper into genes to track infectious disease outbreaks
- Projektart: Marie Skłodowska-Curie Individual Fellowship
- Laufzeit: 1. September 2020 – 31. August 2022 (24 Monate)
- Beitrag für die ETH Zürich: 203’149 €







